En atención a la emergencia en curso causada por el SARS-CoV-2 se presenta una
caracterización de la epidemia en Ecuador. Se aplicó una metodología sugerida por el R
Epidemics Consortium RECON que permite estimar
tempranamente una variable clave como lo es el ritmo reproductivo; dicha metodología
requiere conocer ciertas características del virus, y poseer datos de la incidencia
diaria. Adicionalmente se ha utilizado library(tidyverse).
Este artículo fue actualizado por última vez el 23 de abril, 1 p.m.
Definiciones
Para aplicar la presente metodología es necesario familiarizarse con las siguientes definiciones (Fine 2003), relacionadas con la transmisión de un virus:
El ritmo reproductivo básico (Basic Reproduction Number, R0) es la media de transmisiones por caso; es decir, la cantidad esperada de casos nuevos que un determinado caso generará.
El intervalo serial (Serial Interval, SI) es el tiempo transcurrido entre la aparición de síntomas en dos casos sucesivos; comúnmente se caracteriza al SI a través de su media y desviación estándar en días; no se debe confundir SI con:
El periodo de incubación, entre una infección y la aparición de sus síntomas.
El periodo latente, entre una infección y el comienzo del periodo infeccioso.
En el periodo infeccioso, una persona infectada puede causar nuevas infecciones.
Es crucial entender que tanto el R0 como el SI no son constantes biológicas de un virus, pues son afectados por factores internos -como la vulnerabilidad- y externos -como el ambiente, el contacto social o el tratamiento médico- del individuo infectado. El siguiente gráfico permite visualizar la relación existente entre el SI y los periodos mencionados:
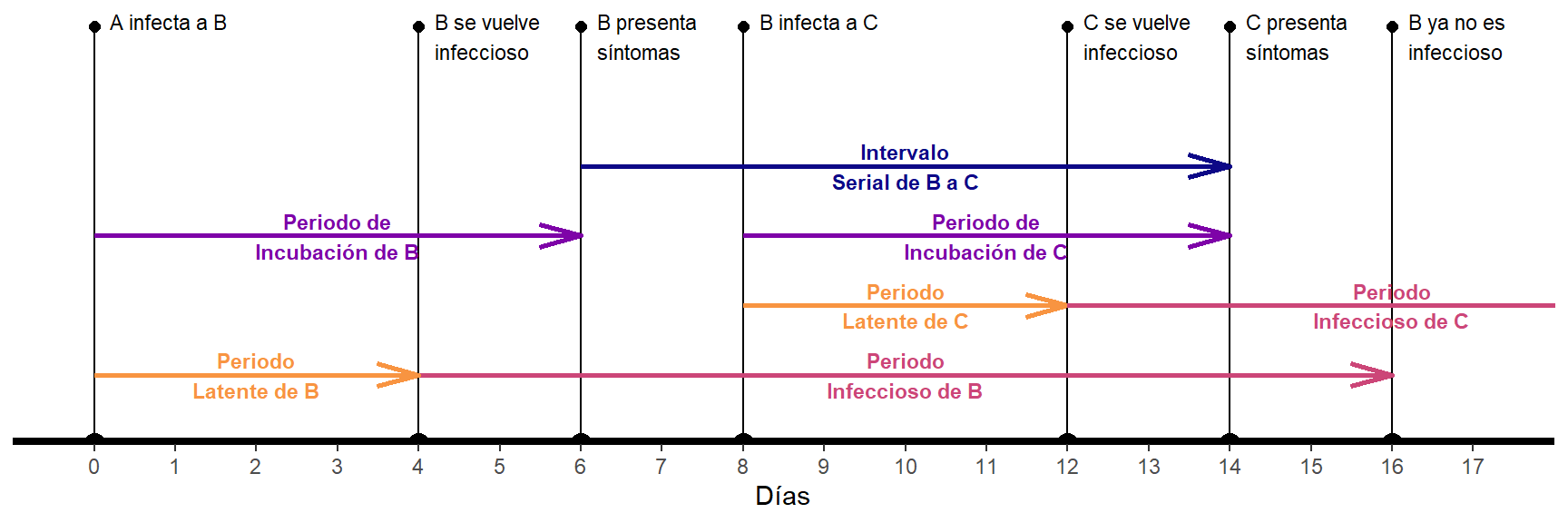
Figure 1: Evento de transmisión con Intervalo Serial positivo
Como lo sugiere el gráfico, el periodo latente puede ser menor que el de incubación, en cuyo caso el individuo infectado se vuelve infeccioso antes de presentar síntomas. En el caso del SARS-CoV-2, se ha observado que en algunos casos el periodo infeccioso empieza un día antes que los síntomas; además, se observó que el periodo de incubación posee una mediana de siete días (Jiang et al. 2020).
Por otra parte, debido a la variabilidad del periodo de incubación, es posible que en una transmisión el individuo infectado presente síntomas antes que el causante de su infección. En este caso el SI se vuelve negativo, tal y como lo presenta el siguiente gráfico:
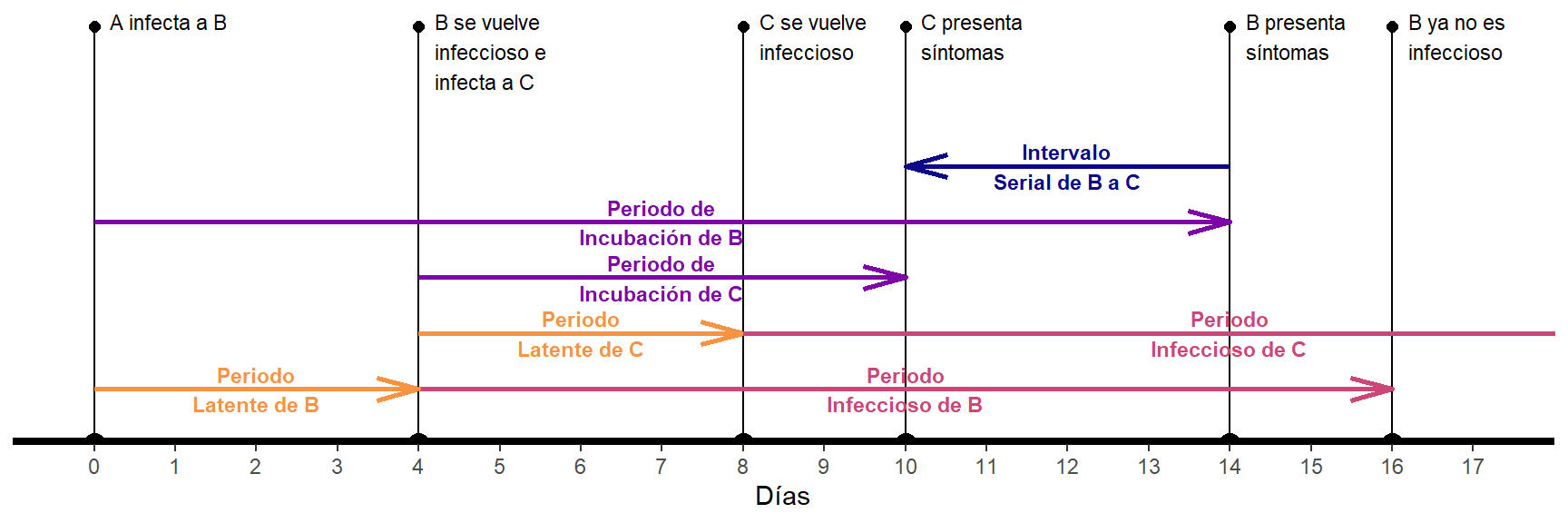
Figure 2: Evento de transmisión con Intervalo Serial negativo
Du et al. (2020) han hallado un porcentaje considerable de SI negativos en eventos de transmisión estudiados. Sin duda la posibilidad de transmitir el virus antes de presentar síntomas -los mismos que pueden ser leves o incluso inexistentes- es un factor que ha contribuido a la propagación descontrolada del SARS-CoV-2.
Obtención de Datos
Los datos requeridos en esta metodología fueron recopilados de infografías publicadas diariamente por el Servicio Nacional de Gestión de Riesgos SNGRE en su cuenta oficial de Twitter. Hasta la fecha (23 de abril), en Ecuador existen 11183 casos confirmados, de los cuales el 5.0076 % han fallecido.
| Infografía | Fecha | Casos Confirmados | Personas Fallecidas |
|---|---|---|---|
| # 59 | 23 de abril | 11183 | 560 |
| # 58 | 22 de abril | 10850 | 537 |
| # 57 | 21 de abril | 10398 | 520 |
| # 56 | 20 de abril | 10128 | 507 |
| # 55 | 19 de abril | 9468 | 474 |
| # 54 | 18 de abril | 9022 | 456 |
| # 53 | 17 de abril | 8450 | 421 |
| # 52 | 16 de abril | 8225 | 403 |
| # 51 | 15 de abril | 7858 | 388 |
| # 50 | 14 de abril | 7603 | 369 |
| # 49 | 13 de abril | 7529 | 355 |
| # 48 | 12 de abril | 7466 | 333 |
| # 47 | 11 de abril | 7257 | 315 |
| # 46 | 10 de abril | 7161 | 297 |
| # 45 | 9 de abril | 4965 | 272 |
| # 44 | 8 de abril | 4450 | 242 |
| # 43 | 7 de abril | 3995 | 220 |
| # 42 | 6 de abril | 3747 | 191 |
| # 41 | 5 de abril | 3646 | 180 |
| # 40 | 4 de abril | 3465 | 172 |
| # 39 | 3 de abril | 3368 | 145 |
| # 38 | 2 de abril | 3163 | 120 |
| # 36 | 1 de abril | 2748 | 93 |
| # 34 | 31 de marzo | 2240 | 75 |
| # 32 | 30 de marzo | 1962 | 60 |
| # 30 | 29 de marzo | 1890 | 57 |
| # 28 | 28 de marzo | 1823 | 48 |
| # 26 | 27 de marzo | 1595 | 36 |
| # 24 | 26 de marzo | 1382 | 34 |
| # 22 | 25 de marzo | 1173 | 28 |
| # 20 | 24 de marzo | 1049 | 27 |
| # 19 | 23 de marzo | 981 | 18 |
| # 18 | 22 de marzo | 789 | 14 |
| # 16 | 21 de marzo | 506 | 7 |
| # 14 | 20 de marzo | 367 | 5 |
| # 12 | 19 de marzo | 199 | 3 |
| # 10 | 18 de marzo | 155 | 2 |
| # 8 | 17 de marzo | 111 | 2 |
| # 6 | 16 de marzo | 58 | 2 |
| # 4 | 15 de marzo | 37 | 2 |
| # 3 | 14 de marzo | 28 | 2 |
| # 1 | 13 de marzo | 23 | 1 |
Ciertos días fueron publicadas dos infografías, en cuyo caso se ha escogido la matutina; los datos completos se encuentran en este enlace. Mientras que la primera infografía corresponde al 13 de marzo, el primer caso ecuatoriano fue anunciado el 29 de febrero; por esto, también se recopilaron comunicados del Ministerio de Salud Pública MSP anteriores a las infografías. Cabe mencionar que, durante dichos comunicados, aún no existían fallecidos por COVID-19 en el país.
| Comunicado | Fecha y Hora de Corte | Casos Confirmados |
|---|---|---|
| link | 12 de marzo, 6 p. m. | 19 |
| link | 10 de marzo, 4 p. m. | 17 |
| link | 8 de marzo, 9 p. m. | 15 |
| link | 6 de marzo, 8 p. m. | 14 |
| link | 5 de marzo, 9 a. m. | 13 |
| link | 4 de marzo, 10 a. m. | 10 |
| link | 2 de marzo, 10 p. m. | 7 |
| link | 1 de marzo, 11 p. m. | 6 |
| link | 29 de febrero, 10 a. m. | 1 |
Modelamiento de Datos
El principal insumo para estimar R0 es la incidencia diaria: la cantidad de casos que presentan síntomas de infección por primera vez (y luego son confirmados) por cada día. Debido a limitaciones de los datos, sin embargo, se ha utilizado en cambio la cantidad de casos confirmados cada día. Esto supone una aproximación a la incidencia pues, desde que un individuo presenta síntomas, hasta que confirman o descartan su infección, puede transcurrir un tiempo considerable. También es cierto que los datos oficiales son limitados, en el sentido de que algunos casos no fueron reportados o fueron asintomáticos.
Ec_data = mutate(COVID19_Salud_Ec, id = NA) %>%
bind_rows(COVID19_Riesgos_Ec[c(1, 3, 4)]) %>%
mutate(day = as.Date(day),
Rate = conf / lag(conf, 1, 0),
Change = conf - lag(conf, 1, 0),
Factor = Change / lag(Change, 1, 0))En el paso anterior se han unido las dos fuentes de datos y se han calculado nuevas variables: la tasa de crecimiento (Rate) es la razón del total de casos confirmados en el país en cierto día, con respecto al día anterior; el cambio (Change) es la diferencia del total, los nuevos casos confirmados cada día; y el factor de crecimiento (Factor) es también una razón, pero del cambio: la división de los nuevos casos confirmados en cierto día, con respecto al día anterior.
Un hecho de las epidemias es que, incluso en el peor escenario, no pueden crecer indefinidamente pues se agotaría la población susceptible al contagio. Cuando la tasa de crecimiento -la cual define un crecimiento exponencial- se establezca definitivamente en su valor mínimo (que es uno), los casos confirmados detendrán su incremento. Por otra parte, resulta útil vigilar los cambios en el factor de crecimiento: valores menores a uno indican una reducción del avance de la epidemia y viceversa, valores mayores a uno indican que el crecimiento exponencial continúa.
En los siguientes gráficos se excluyen los datos previos al 24 de marzo, fecha en que se superó los mil casos; el color representa la tasa de crecimiento, y el multiplicador el factor de crecimiento.
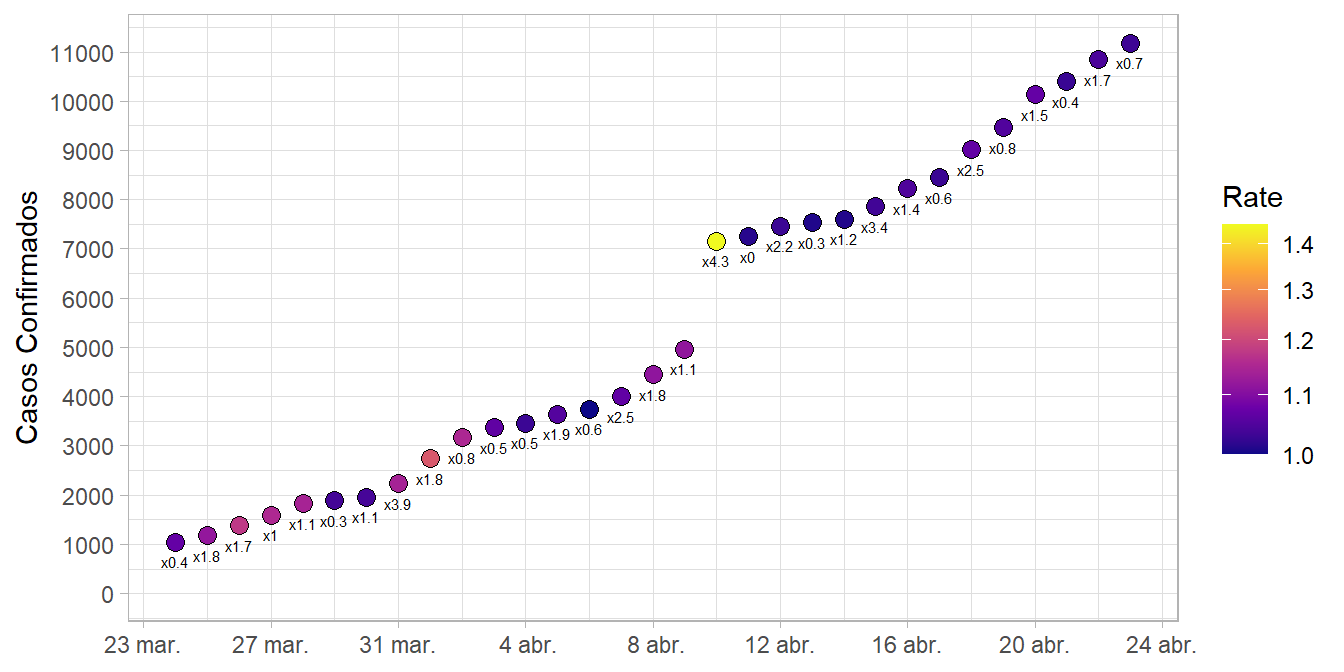
Figure 3: Total de casos COVID-19 confirmados en Ecuador, incluyendo tasas y factores de crecimiento
Ajuste de Regresión Exponencial
Debido a la variación en el cambio del total de casos confirmados, el ajuste de una única regresión exponencial no parece explicar completamente el crecimiento de la epidemia en Ecuador. Al parecer existen tres patrones de crecimiento, con fechas de separación del 22 de marzo y 9 de abril; ambas fechas fueron escogidas de manera que maximicen -en función del coeficiente de determinación- el ajuste de los modelos. Lo que es más importante, la primera fecha es congruente con las medidas de control adoptadas el 17 de marzo -una semana antes del 23 de marzo- en Ecuador, que incluyen: toque de queda, prohibición de reuniones sociales, restricción del tránsito vehicular, entre otros.
mod1 = lm(log(conf) ~ day, Ec_data[10:19, ]) # hasta el 22 de marzo
mod2 = lm(log(conf) ~ day, Ec_data[20:37, ]) # del 23 de marzo al 9 de abril
mod3 = lm(log(conf) ~ day, Ec_data[-{1:37}, ]) # desde el 10 de abril| Regresion | Inicio | Fin | Coeficiente | Pendiente | Crecimiento |
|---|---|---|---|---|---|
| 1° | 13 de marzo | 22 de marzo | 0.9919 | 0.4096 | 50.62 % |
| 2° | 23 de marzo | 9 de abril | 0.9774 | 0.0956 | 10.03 % |
| 3° | 10 de abril | … | 0.9636 | 0.0370 | 3.76 % |
En función de la pendiente del modelo exponencial se calcula la tasa de crecimiento más ajustada a la realidad; de esta manera se halló que, antes de surtir efecto las medidas, los casos confirmados crecieron un 50.6201 % cada día. Después de las medidas se observó un crecimiento del 10.0287 % diario; finalmente y hasta la fecha, el crecimiento es del 3.7646 %. A continuación se presenta en dos gráficos los modelos ajustados; la versión de la derecha, al poseer una escala logarítmica, permite apreciar con mayor claridad la diferencia en el patrón de crecimiento.
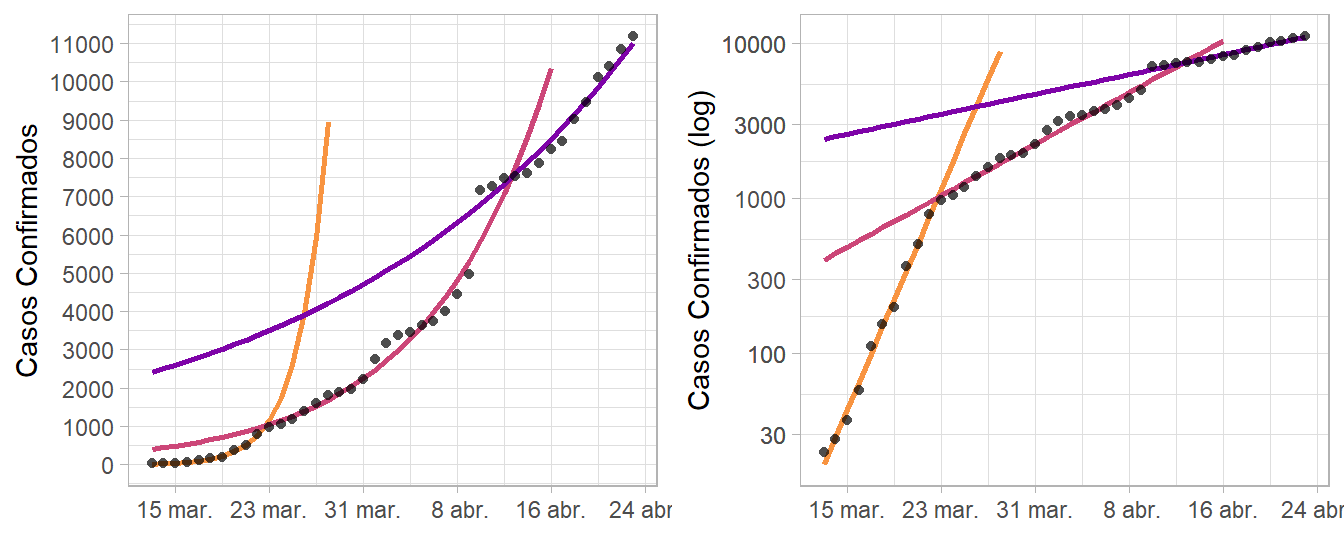
Figure 4: Primera (naranja), segunda (rosada) y tercera (morada) regresión exponencial ajustada
Estimación del Ritmo Reproductivo
Independientemente de haber hallado un buen ajuste a través de tres regresiones, la
metodología requiere estimar un único R0 utilizando incidence(); es importante
introducir en esta función todos los datos disponibles, incluso si existen días sin
datos (como ocurre en comunicados del MSP).
El objeto generado por incidence() contiene la incidencia diaria necesaria para la
estimación; no obstante, hace falta conocer el SI característico de la epidemia. En
este contexto, lo recomendable es obtener la media y desviación estándar del SI a
partir de la literatura; una estimación temprana es recomendable, pues utilizar los
datos de una epidemia avanzada incluiría los efectos de las medidas de control,
subestimando de esta manera el SI (Fine 2003).
Se ha considerado dos artículos recientes que calculan el SI del SARS-CoV-2. Nishiura, Linton, and Akhmetzhanov (2020) han obtenido una media de 4.7 y una desviación estándar de 2.9 días a partir de 28 casos, ajustándose a una distribución lognormal. Se trata de un SI notablemente más corto que el otros coronavirus (se ha calculado para el SARS, por ejemplo, una media de 8.4 días), lo cual indica una velocidad superior de transmisión. Por otra parte, Du et al. (2020) usaron 468 casos en sus cálculos, 59 de ellos con SI negativo (como el ejemplificado en el gráfico 2); debido a los valores negativos, la distribución normal generó el mejor ajuste, con una media de 3.96 y una desviación estándar de 4.75 días.
Con el propósito de escoger una distribución del SI, se ha iterado el cálculo de R0
-con ayuda de la función get_R() aquí definida- variando la fecha final de los datos
introducidos, y utilizando cada vez las distribuciones sugeridas en los dos artículos.
library(earlyR)
my_getr = function(i, mn = 3.96, sd = 4.75){
if(i < 14) return(NA)
ml = get_R(Ec_inci[1:i], si_mean = mn, si_sd = sd, days = 0)
if(length(ml$R_ml) == 1) return(ml$R_ml) else return(0)
}
Ec_data = Ec_data %>% mutate(
Nishiura = map_dbl(4 + row_number(), my_getr, mn = 4.7, sd = 2.9),
Du_et_al = map_dbl(4 + row_number(), my_getr))Figure 5: Ritmo reproductivo estimado según distribuciones SI de dos artículos, incluyendo tasas de crecimiento
En este gráfico R0 = 0 quiere decir que falló el cálculo de esta variable, lo cual sucede al introducir todos los datos hasta el 3 de abril (y en adelante). Es evidente que, independientemente del artículo considerado, R0 incrementa junto con la tasa de crecimiento; de manera que la estimación depende mucho de los datos disponibles. Adicionalmente, si bien la diferencia no es grande, la estimación de acuerdo a Du et al. (2020) siempre es mayor.
En este punto hay que reconocer que se ignora si la metodología RECON solo admite distribuciones positivas, como la lognormal. En consecuencia, se decidió utilizar la distribución según Nishiura, Linton, and Akhmetzhanov (2020) y los datos hasta el 26 de marzo, presentándose alrededor de esta fecha la mayor estabilidad en los R0 estimados. De esta manera, se obtuvo un intervalo de confianza (CI) al 95 % de 1.66 a 1.84.
R_get = get_R(Ec_inci[1:27], si_mean = 4.7, si_sd = 2.9, days = 0)
R_sam = sample_R(R_get, 1e4); quantile(R_sam, c(.025, .5, .975))## 2.5% 50% 97.5%
## 1.662 1.752 1.842Proyecciones
La función project() permite utilizar una muestra de valores R0 probables y proyectar
el número esperado de casos confirmados en los próximos días. A continuación se proponen
tres escenarios diferentes, reduciendo cada vez el valor R0 más probable; cada escenario
se proyecta a partir del 27 de marzo (dado que R0 fue estimado hasta el 26). En los
gráficos, la región sombreada representa el 95 % CI de la proyección.
Primer escenario: R0 al 100 %
De acuerdo a la estimación de R0 su valor más probable es 1.7518 lo cual quiere decir que, cada cuatro individuos infectados, se han infectado siete más en promedio. El cálculo de la proyección y su CI se realiza como sigue:
library(projections)
Ec_proj = project(Ec_inci[1:27], R_sam, R_get$si, n_days = 13) # hasta 8 de abril
Ec_data = as_tibble(t(as.matrix(Ec_proj))) %>%
summarise(across(.fns = list(
pro.low = ~quantile(., .025),
pro.hig = ~quantile(., .975),
pro.med = median))) %>%
pivot_longer(everything(), names_to = c("day", "name"), names_sep = "_") %>%
pivot_wider() %>%
mutate(across(where(is.numeric), ~cumsum(.) + 1382), across(day, as.Date)) %>%
full_join(Ec_data, .)Asumir que R0 mantiene su valor más probable al 100 % representa un escenario donde ninguna medida fue introducida para contener la epidemia. Ciertamente este no es el caso, razón por la cual no se ha proyectado este escenario más allá del 8 de abril, fecha en la cual hubiesen existido ya nueve mil casos confirmados.
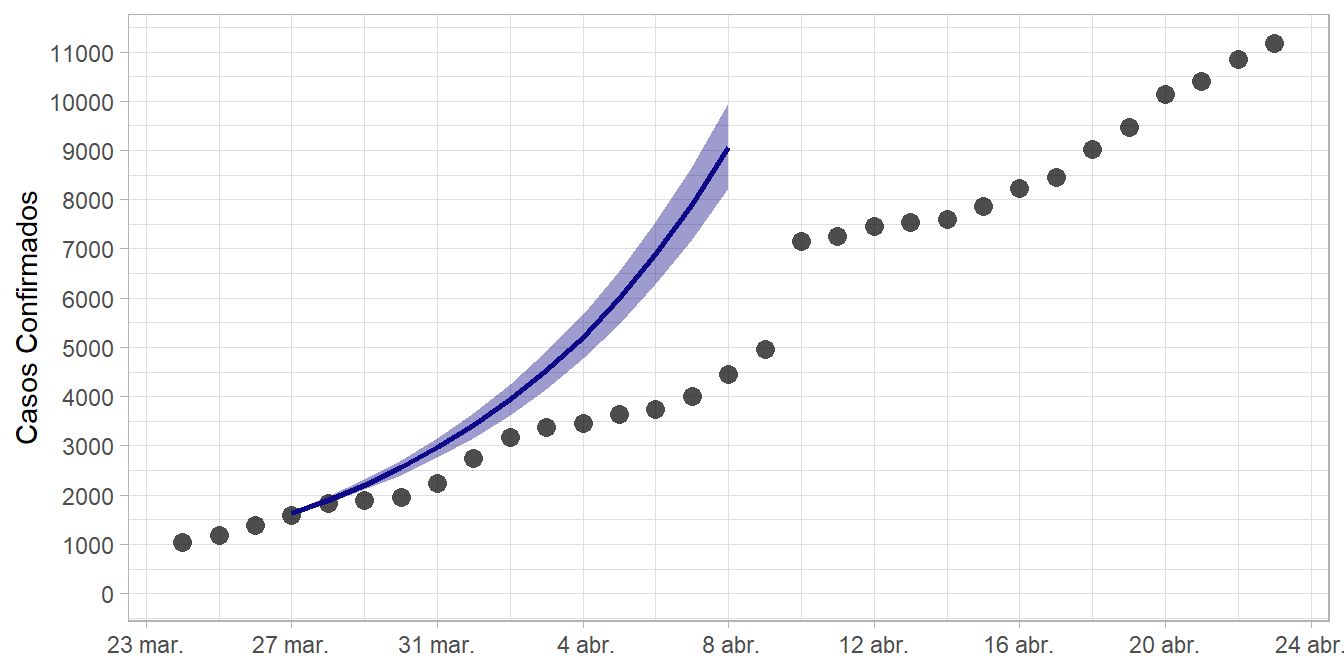
Figure 6: Proyección de casos COVID-19 confirmados en Ecuador, si R0 = 1.75
Segundo escenario: R0 al 75 %
Supongamos que, a través de las medidas adoptadas en Ecuador, el ritmo reproductivo se redujo progresivamente hasta alcanzar el 75 % del valor más probable original, esto es 1.3138. Significaría que, cada tres individuos infectados, habría aproximadamente cuatro casos nuevos. De acuerdo al gráfico, parecería ser el escenario más cercano a la realidad hasta ahora; si fuera así, el 1 de mayo se alcanzaría los 24 mil casos confirmados.
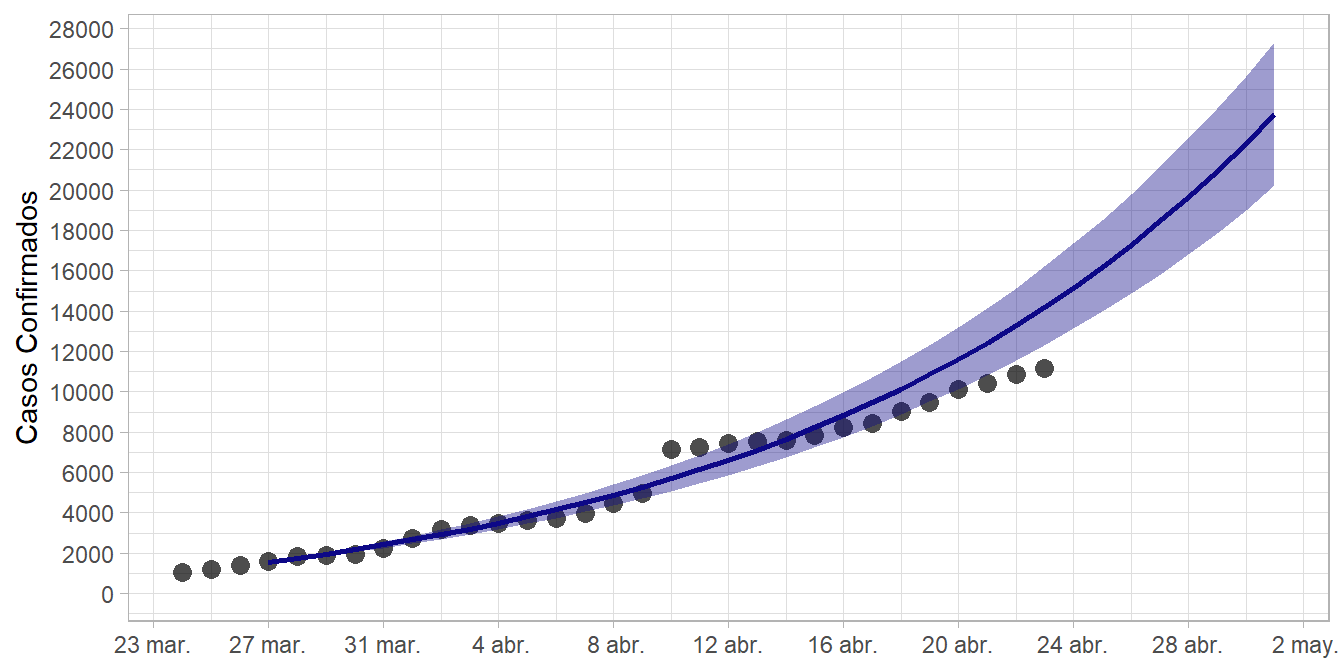
Figure 7: Proyección de casos COVID-19 confirmados en Ecuador, si R0 = 1.31
Tercer escenario: R0 al 50 %
Supongamos finalmente que R0 se redujo a la mitad de su valor más probable original, es decir a 0.8759. Cada ocho individuos infectados habría aproximadamente siete casos nuevos, significando una reducción del avance de la epidemia. No obstante esto es una subestimación del escenario real, pues hasta la fecha se habrían confirmado solamente unos 3800 casos.
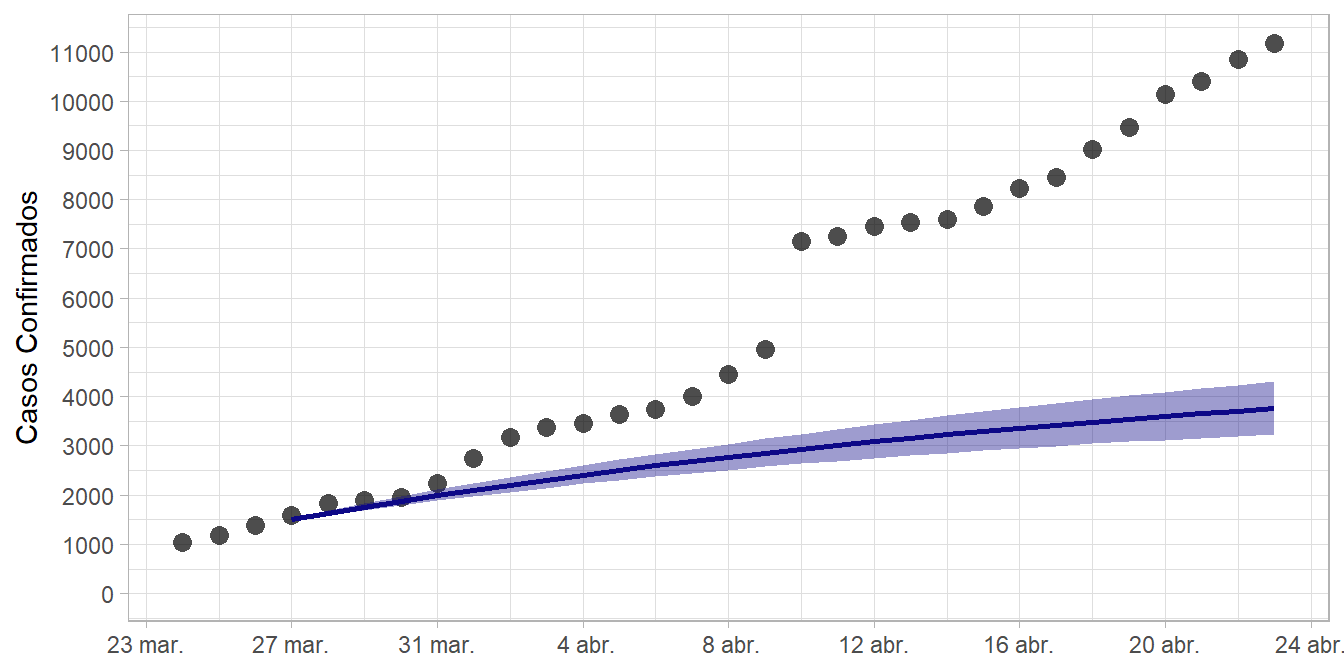
Figure 8: Proyección de casos COVID-19 confirmados en Ecuador, si R0 = 0.88
Conclusiones
Este análisis consideró lo que la literatura sobre el virus SARS-CoV-2 sugiere hasta el momento. Si bien más investigaciones se encuentran en camino, los resultados ya han demostrado la magnitud de la amenaza que enfrentamos; recordemos que la transmisión es más rápida comparada con otros coronavirus, y puede pasar inadvertida -si ocurre antes que el inicio de síntomas- en muchos casos.
Una desventaja de esta caracterización es que utilizó estimaciones -concretamente la distribución del intervalo serial- realizadas en un contexto distinto. Como se mencionó en las definiciones, estas características epidemiológicas dependen de factores que varían entre sociedades y escenarios de control; hace falta una estimación del SI con los datos observados en Ecuador. Con todo, el valor R0 obtenido es congruente con lo observado en varios países alrededor del mundo, y el consenso parece ser que la epidemia del COVID-19 posee un ritmo reproductivo entre uno y dos. (Du et al. 2020) han calculado R0 = 1.32 para casos chinos fuera de Wuhan. Con el control adoptado en Ecuador, el ritmo reproductivo parece haber decrecido entre un 25 (gráfico 7) y un 50 %.
Como conclusión las proyecciones aquí presentadas son exploratorias; en vez de predecir lo que sucederá en los próximos días, se pretende explicar cuánto se ha reducido el avance de la epidemia hasta ahora. Sabemos que si R0 < 1 los casos confirmados detendrán su crecimiento exponencial (gráfico 4), lo cual es crucial para no colapsar los servicios de salud. También podríamos discernir si el crecimiento dejará de ser exponencial, observando cuándo los factores de crecimiento (gráfico 3) son menores que uno. Por último, aunque no se lo ha hecho aquí, mencionar que es posible generar un modelo compartimental SIR, para analizar cuándo ocurrirá el pico de individuos infectados.
Du, Zhanwei, Lin Wang, Xiaoke Xu, Ye Wu, Benjamin Cowling, and Lauren Meyers. 2020. “Serial Interval of Covid-19 Among Publicly Reported Confirmed Cases.” Emerging Infectious Diseases. https://doi.org/10.3201/eid2606.200357.
Fine, Paul. 2003. “The Interval Between Successive Cases of an Infectious Disease.” American Journal of Epidemiology 158 (December): 1039–47. https://doi.org/10.1093/aje/kwg251.
Jiang, Xue, Yawei Niu, Xiong Li, Lin Li, Wenxiang Cai, Yucan Chen, Bo Liao, and Edwin Wang. 2020. “Is a 14-Day Quarantine Period Optimal for Effectively Controlling Coronavirus Disease 2019 (Covid-19)? (PREPRINT).” medRxiv. https://doi.org/10.1101/2020.03.15.20036533.
Nishiura, Hiroshi, Natalie Linton, and Andrei Akhmetzhanov. 2020. “Serial Interval of Novel Coronavirus (Covid-19) Infections.” International Journal of Infectious Diseases 93 (April): 284–86. https://doi.org/10.1016/j.ijid.2020.02.060.
